Ensemble de données sur les tumeurs cérébrales
Un ensemble de données de détection de tumeurs cérébrales est constitué d'images médicales provenant d'IRM ou de scanners, contenant des informations sur la présence, l'emplacement et les caractéristiques des tumeurs cérébrales. Cet ensemble de données est essentiel pour entraîner des algorithmes de vision par ordinateur afin d'automatiser l'identification des tumeurs cérébrales, ce qui contribue au diagnostic précoce et à la planification du traitement dans les applications de soins de santé.
Regarder : Détection de tumeurs cérébrales avec la plateforme Ultralytics
Structure du jeu de données
Le jeu de données des tumeurs cérébrales est divisé en deux sous-ensembles :
- Ensemble d'entraînement : Composé de 893 images, chacune accompagnée des annotations correspondantes.
- Ensemble de test : Comprenant 223 images, avec des annotations appariées pour chacune.
L'ensemble de données contient deux classes :
- Négatif: Images sans tumeurs cérébrales
- Positif : Images avec des tumeurs cérébrales
Applications
L'application de la détection des tumeurs cérébrales à l'aide de la vision par ordinateur permet le diagnostic précoce, la planification du traitement et le suivi de la progression de la tumeur. En analysant les données d'imagerie médicale telles que les IRM ou les tomodensitométries, les systèmes de vision par ordinateur aident à identifier avec précision les tumeurs cérébrales, contribuant ainsi à une intervention médicale rapide et à des stratégies de traitement personnalisées.
Les professionnels de la santé peuvent exploiter cette technologie pour :
- Réduire le temps de diagnostic et améliorer la précision
- Aider à la planification chirurgicale en localisant précisément les tumeurs
- Surveiller l'efficacité du traitement au fil du temps
- Soutien à la recherche en oncologie et en neurologie
YAML du jeu de données
Un fichier YAML (Yet Another Markup Language) est utilisé pour définir la configuration de l'ensemble de données. Il contient des informations sur les chemins d'accès, les classes et d'autres informations pertinentes de l'ensemble de données. Dans le cas de l'ensemble de données sur les tumeurs cérébrales, le brain-tumor.yaml fichier est maintenu à l'adresse https://github.com/ultralytics/ultralytics/blob/main/ultralytics/cfg/datasets/brain-tumor.yaml.
ultralytics/cfg/datasets/brain-tumor.yaml
# Ultralytics 🚀 AGPL-3.0 License - https://ultralytics.com/license
# Brain-tumor dataset by Ultralytics
# Documentation: https://docs.ultralytics.com/datasets/detect/brain-tumor/
# Example usage: yolo train data=brain-tumor.yaml
# parent
# ├── ultralytics
# └── datasets
# └── brain-tumor ← downloads here (4.21 MB)
# Train/val/test sets as 1) dir: path/to/imgs, 2) file: path/to/imgs.txt, or 3) list: [path/to/imgs1, path/to/imgs2, ..]
path: brain-tumor # dataset root dir
train: images/train # train images (relative to 'path') 893 images
val: images/val # val images (relative to 'path') 223 images
# Classes
names:
0: negative
1: positive
# Download script/URL (optional)
download: https://github.com/ultralytics/assets/releases/download/v0.0.0/brain-tumor.zip
Utilisation
Pour entraîner un modèle YOLO26 sur l'ensemble de données des tumeurs cérébrales pendant 100 époques avec une taille d'image de 640, utilisez les extraits de code fournis. Pour une liste détaillée des arguments disponibles, consultez la page Entraînement du modèle.
Exemple d'entraînement
from ultralytics import YOLO
# Load a model
model = YOLO("yolo26n.pt") # load a pretrained model (recommended for training)
# Train the model
results = model.train(data="brain-tumor.yaml", epochs=100, imgsz=640)
# Start training from a pretrained *.pt model
yolo detect train data=brain-tumor.yaml model=yolo26n.pt epochs=100 imgsz=640
Exemple d'inférence
from ultralytics import YOLO
# Load a model
model = YOLO("path/to/best.pt") # load a brain-tumor fine-tuned model
# Inference using the model
results = model.predict("https://ultralytics.com/assets/brain-tumor-sample.jpg")
# Start prediction with a finetuned *.pt model
yolo detect predict model='path/to/best.pt' imgsz=640 source="https://ultralytics.com/assets/brain-tumor-sample.jpg"
Images et annotations d'exemple
L'ensemble de données sur les tumeurs cérébrales comprend un large éventail d'images médicales présentant des scans cérébraux avec et sans tumeurs. Vous trouverez ci-dessous des exemples d'images de l'ensemble de données, accompagnées de leurs annotations respectives.
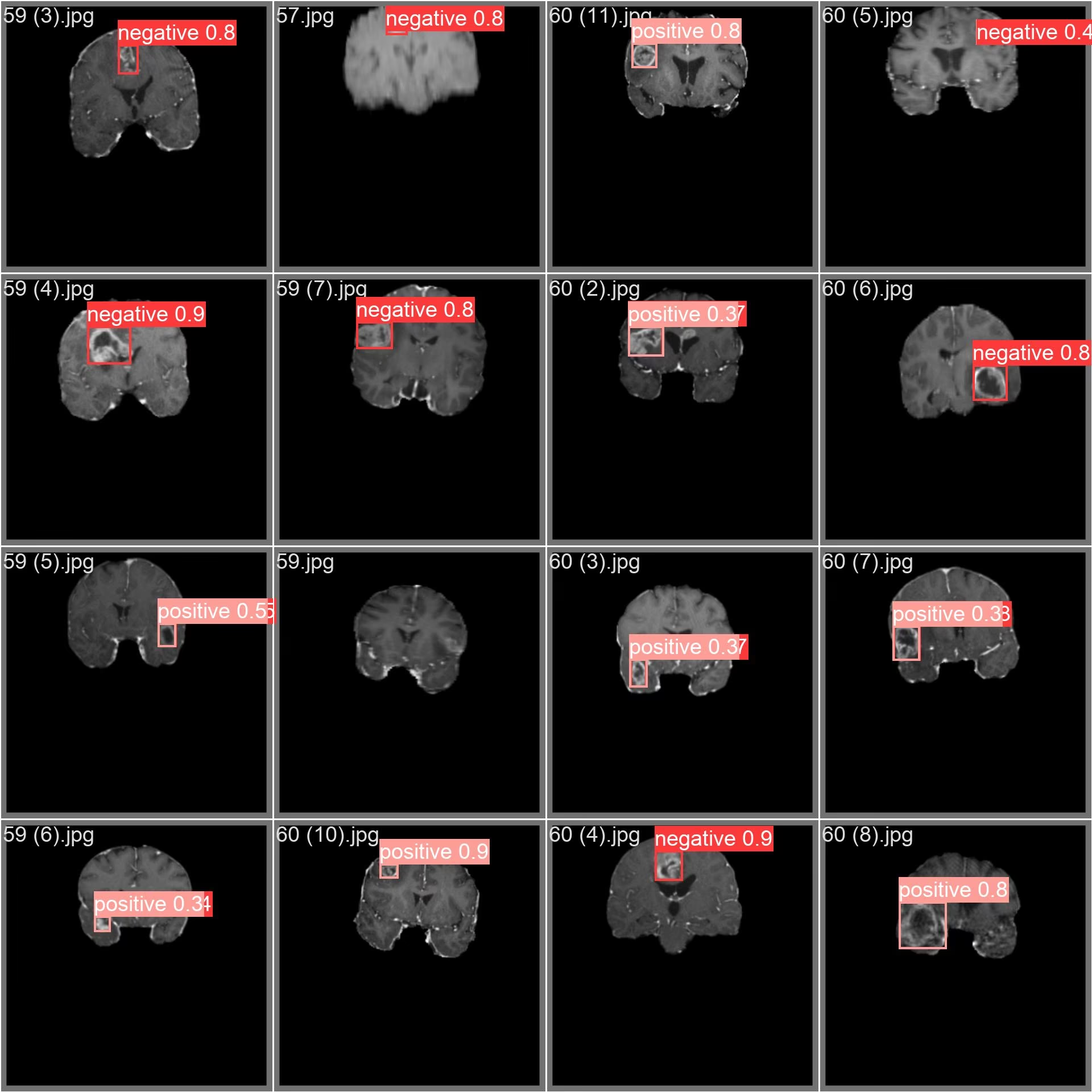
- Image mosaiquée : Une série d’images de l’ensemble de données mosaiquées utilisées pour l’entraînement est affichée ici. La mosaique, une technique d’entraînement, regroupe plusieurs images en une seule, améliorant ainsi la diversité des lots. Cette approche contribue à améliorer la capacité du modèle à généraliser différentes tailles, formes et emplacements de tumeurs dans les scans cérébraux.
Cet exemple met en évidence la diversité et la complexité des images dans l'ensemble de données de tumeurs cérébrales, soulignant les avantages de l'incorporation de la mosaïque pendant la phase d'entraînement pour l'analyse d'images médicales.
Citations et remerciements
L'ensemble de données a été mis à disposition sous la licence AGPL-3.0.
Si vous utilisez cet ensemble de données dans vos travaux de recherche ou de développement, veuillez le citer de manière appropriée :
@dataset{Ultralytics_Brain_Tumor_Dataset_2023,
author = {Ultralytics},
title = {Brain Tumor Detection Dataset},
year = {2023},
publisher = {Ultralytics},
url = {https://docs.ultralytics.com/datasets/detect/brain-tumor/}
}
FAQ
Quelle est la structure de l'ensemble de données sur les tumeurs cérébrales disponible dans la documentation Ultralytics ?
Le jeu de données sur les tumeurs cérébrales est divisé en deux sous-ensembles : l’ensemble d’entraînement comprend 893 images avec les annotations correspondantes, tandis que l’ensemble de test comprend 223 images avec des annotations appariées. Cette division structurée aide à développer des modèles de vision par ordinateur robustes et précis pour la détection des tumeurs cérébrales. Pour plus d’informations sur la structure de l’ensemble de données, consultez la section Structure de l’ensemble de données.
Comment puis-je entraîner un modèle YOLO26 sur l'ensemble de données des tumeurs cérébrales à l'aide d'Ultralytics ?
Vous pouvez entraîner un modèle YOLO26 sur l'ensemble de données des tumeurs cérébrales pendant 100 époques avec une taille d'image de 640px en utilisant les méthodes Python et CLI. Vous trouverez ci-dessous les exemples pour les deux :
Exemple d'entraînement
from ultralytics import YOLO
# Load a model
model = YOLO("yolo26n.pt") # load a pretrained model (recommended for training)
# Train the model
results = model.train(data="brain-tumor.yaml", epochs=100, imgsz=640)
# Start training from a pretrained *.pt model
yolo detect train data=brain-tumor.yaml model=yolo26n.pt epochs=100 imgsz=640
Pour obtenir une liste détaillée des arguments disponibles, consultez la page Formation.
Quels sont les avantages de l'utilisation de l'ensemble de données sur les tumeurs cérébrales pour l'IA dans le domaine de la santé ?
L'utilisation de l'ensemble de données sur les tumeurs cérébrales dans les projets d'IA permet un diagnostic précoce et une planification du traitement des tumeurs cérébrales. Elle facilite l'automatisation de l'identification des tumeurs cérébrales grâce à la vision par ordinateur, ce qui permet des interventions médicales précises et rapides, et soutient des stratégies de traitement personnalisées. Cette application recèle un potentiel important pour améliorer les résultats pour les patients et l'efficacité médicale. Pour plus d'informations sur les applications de l'IA dans le domaine de la santé, consultez les solutions de santé d'Ultralytics.
Comment effectuer une inférence à l'aide d'un modèle YOLO26 affiné sur l'ensemble de données des tumeurs cérébrales ?
L'inférence utilisant un modèle YOLO26 affiné peut être effectuée avec des approches Python ou CLI. Voici les exemples :
Exemple d'inférence
from ultralytics import YOLO
# Load a model
model = YOLO("path/to/best.pt") # load a brain-tumor fine-tuned model
# Inference using the model
results = model.predict("https://ultralytics.com/assets/brain-tumor-sample.jpg")
# Start prediction with a finetuned *.pt model
yolo detect predict model='path/to/best.pt' imgsz=640 source="https://ultralytics.com/assets/brain-tumor-sample.jpg"
Où puis-je trouver la configuration YAML pour l'ensemble de données sur les tumeurs cérébrales ?
Le fichier de configuration YAML pour l'ensemble de données des tumeurs cérébrales se trouve à l'adresse brain-tumor.yaml. Ce fichier comprend les chemins d'accès, les classes et d'autres informations pertinentes nécessaires à l'entraînement et à l'évaluation des modèles sur cet ensemble de données.